今日の実験結果がNegativeだったので早く帰って来た管理人です😂
今朝Xで言及していた論文が個人的にどストライクだったので、もう紹介記事を作ってしまおうと思います。
この論文はMITでRibosome profilingやCRISPR screeiningの開発を主導した、Jonathan Weissmanラボからの論文です。
どちらも現在の生命科学の研究では超重要な技術となっており、Commodity化する汎用性と、今回のlocal ribosome profilingやin vivo CRISPR screeningのような拡張性を備えたバケモノ技術です。
今回はこのRibosome profilingという技術を発展させて、ミトコンドリアの外膜上でリボソームがミトコンドリアタンパクを局所翻訳していることを証明した論文です。
タイトルは「Proximity-specific ribosome profiling reveals the logic of localized mitochondrial translation」になります(https://www.cell.com/cell/fulltext/S0092-8674(25)00916-X)。
めちゃめちゃありがたいことにCC-BYになっているので、画像付きで紹介できます!こんな良質な論文が著作権を気にせず紹介できるのは本当に嬉しいですね(https://s100.copyright.com/AppDispatchServlet?publisherName=ELS&contentID=S009286742500916X&orderBeanReset=true&orderSource=Phoenix)。
ミトコンドリアにおける局所翻訳の最初の証明
古典的にはミトコンドリアに存在するタンパク質は2種類に分けられます
・一つはミトコンドリアDNAにコードされて、そのままミトコンドリア内で翻訳されるタンパク質
・もう一つは核にコードされて細胞質で翻訳された後、ミトコンドリアに輸送されるタンパク質です
しかし最近これ以外の翻訳経路も存在することが示唆されるようになっており、酵母(Saccharomyces cerevisiae)では結構前からRNAがミトコンドリア上で局所翻訳されていることが示されていたようです。
Weissmanラボも2014年にRibosome profilingを応用して、この酵母で局所翻訳が起きていることを証明しScienceに報告しています(https://www.science.org/doi/10.1126/science.1257522)。
しかし哺乳類では酵母で同定した局所翻訳に関わる遺伝子が保存されておらず、また技術的な制約から2014年の手法が応用出来なかったことで、直接的な証拠がつかめていませんでした。
ちなみに、Ribosomeがミトコンドリア外膜上にあることや、mRNAがリボソーム外膜近くにあることは電子顕微鏡やAPEX-seqで明らかになっていました。しかし、実際に翻訳されているかは証明されていませんでした。
APEX-seq: https://www.sciencedirect.com/science/article/pii/S0092867419305550
瞬間を切り取る技術の開発!
Ribosome profilingはリボソーム内部に存在して実際に翻訳されているリードだけを回収し、RNAの翻訳効率を網羅的に評価することができる手法です。日本だと東大の岩崎先生が有名ですね。
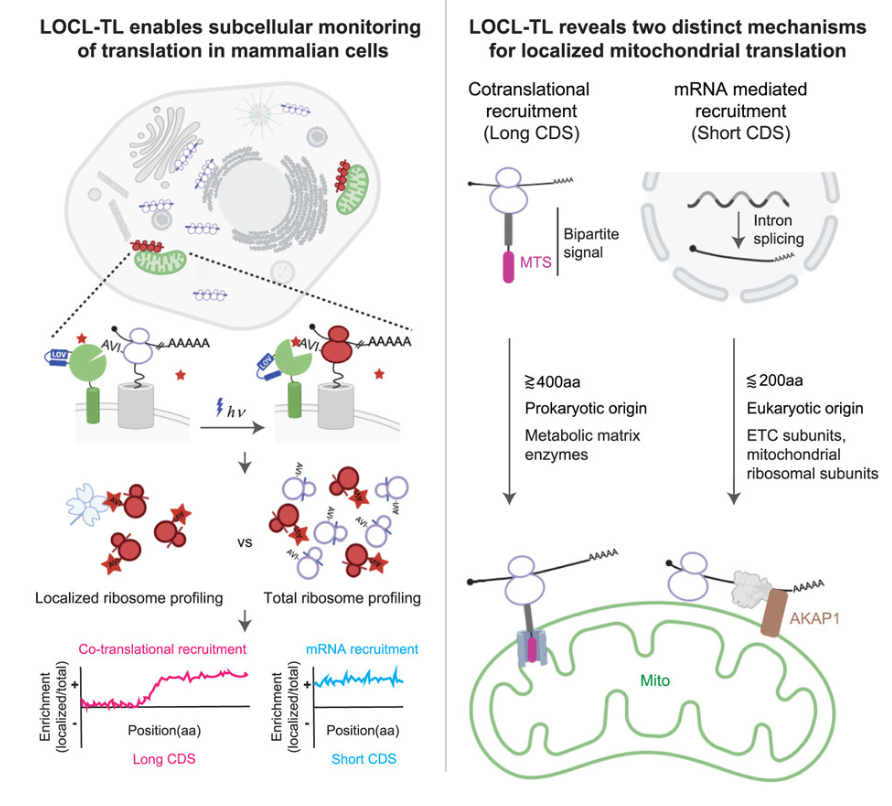
局所翻訳を評価するために、彼らは対象の小器官上にビオチンリガーゼ(BirA)を発現させ、リボソーム側にAvi-tagを発現させることで、小器官にリボソームが近接した時にリボソームがビオチン化されるようにしています。その後ビオチン化されたRibosomeのみをStreptavidinで回収し、Ribosome profilingにかけています。
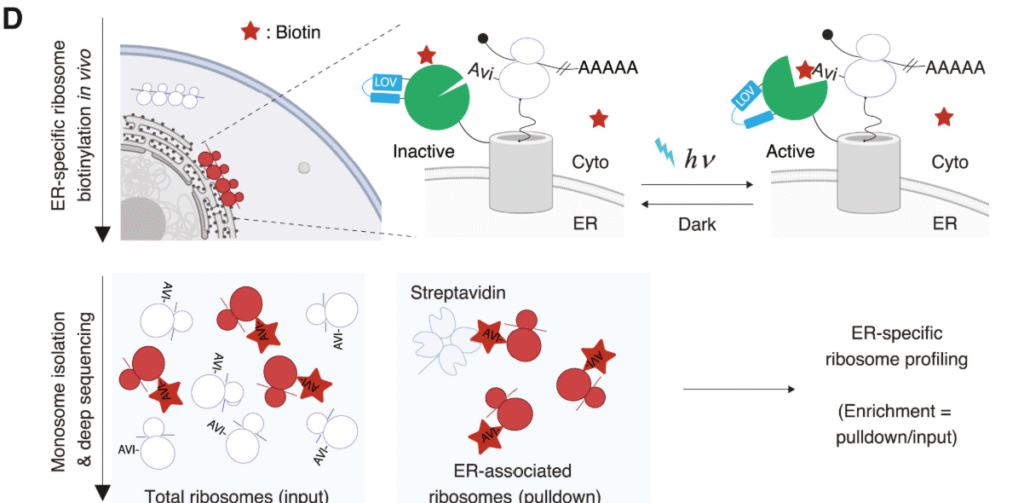
この時に問題になるのが、翻訳を終えたリボソームはRNAから離れて細胞質の別のRNAを翻訳してしまうことです。そのため、ゆっくりしているとビオチン化されたリボソームがどんどん細胞質でも増えてしまいます。
この問題を解決するために彼らはブルーライトを当てたときだけ、ビオチンリガーゼが活性化するLOV-BirAを開発しました。こうすることで10分間光を当ててる間だけ、小器官上のリボソームがビオチン化されます。
この技術をまずは膜上での翻訳がすでに知られている小胞体で行ったところ、分泌されることが知られているタンパクが下図のようにエンリッチされており、Workすることを確認できました。
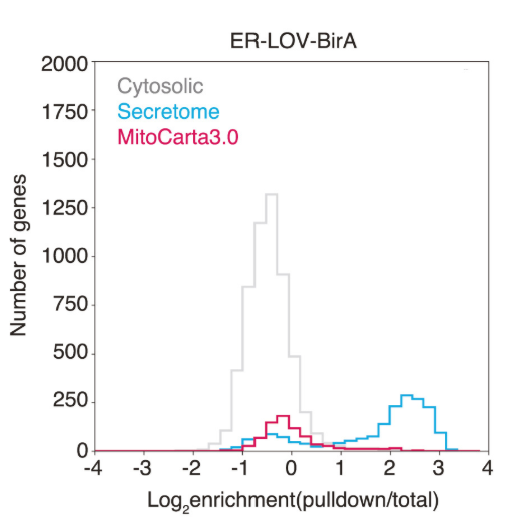
ミトコンドリア上における局所翻訳の謎
次に目的のミトコンドリアの膜上のタンパクを色々試して、OMP25の膜貫通ドメインにLOV-BirをFusionすると、核にコードされるミトコンドリアタンパクの翻訳を捉えることが出来ることが分かりました。
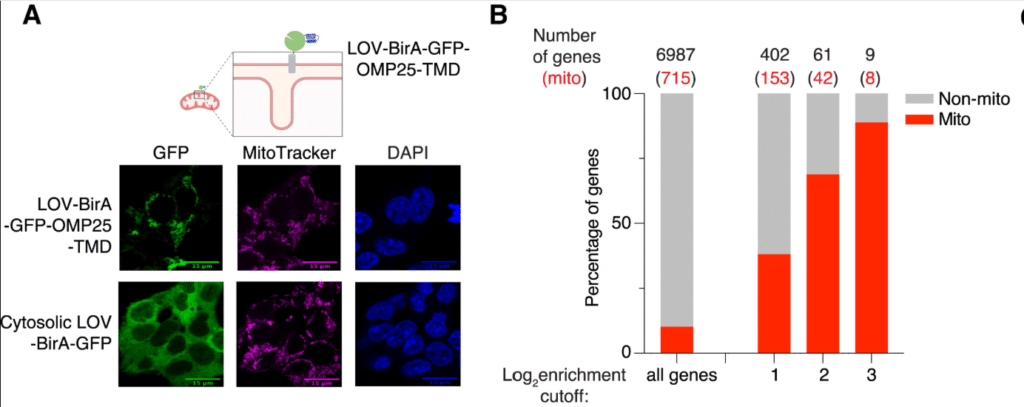
ここで彼らは面白いことに気が付きます。
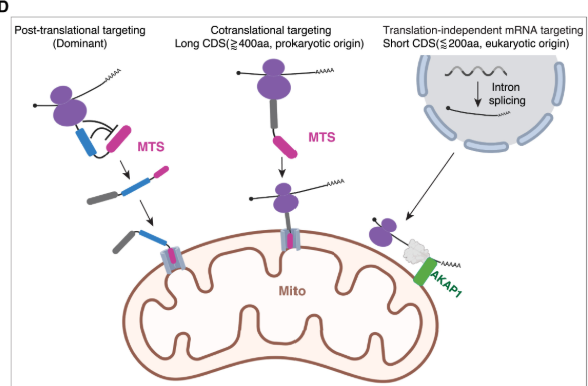
核にコードされるミトコンドリアタンパクのうちで、局所翻訳されるものはCDSがある程度長いものと短いものに分離したのです。
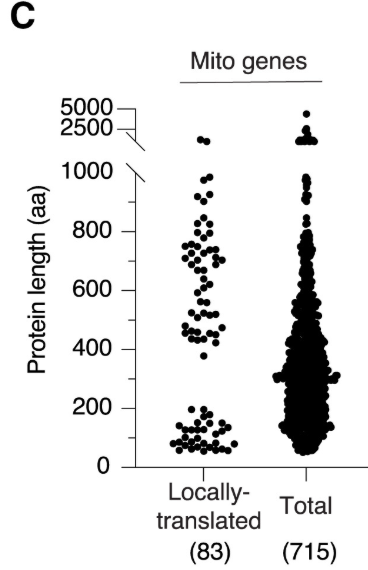
CDSが長い遺伝子と短い遺伝子を比較すると、下記のような傾向がありました。
長い遺伝子:アミノアシルtRNA合成酵素や代謝経路に関わる、マトリックスに局在する
短い遺伝子:電子伝達系やミトコンドリアリボソームに関わる、内膜に局在する
ここで彼らはこの2群のmRNAは別のメカニズムで外膜上にリクルートされるのではないかと仮説を立てました。そこでビオチン化されたリボソームに含まれるリードを確認すると、短いmRNAでは初めからリードが存在して、長いRNAでは200塩基ほど進んだところからリボソームがビオチン化されることが分かりました。
つまり短いRNAはもともと外膜上にリクルートされていて最初から外膜上で翻訳され、長いRNAは小胞体での局所翻訳と同様に細胞質で途中まで翻訳されてからリクルートされていることを意味しています。
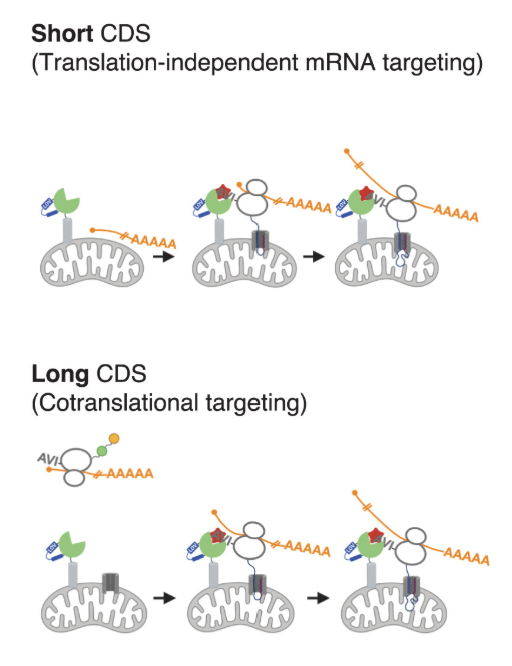
ふたつのメカニズム解明をめざして
まず彼らは長いmRNAがリクルートされるメカニズムを調べます。
長いmRNAのリボソームがビオチン化するタイミングを調べると、CDSの長さに関わらず、共通して翻訳開始から250アミノ酸ほど進んだところでリクルートされることが分かりました。
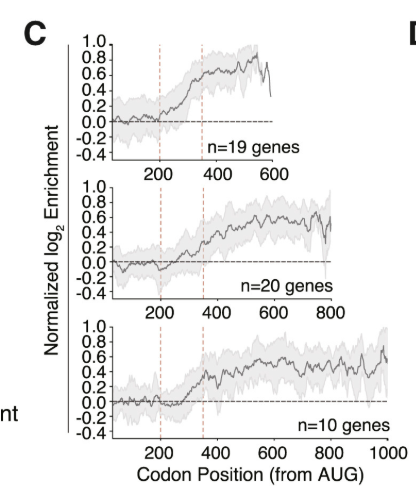
彼らはここから変異タンパクを色々作って、これらのRNAに含まれるミトコンドリア標的シグナル(MTS)の下流200塩基ほどに位置する配列がリクルートに重要だろうと推測しています。この配列は、フレームシフト変異でもリクルートが影響を受けたことから、RNAの配列によるものというよりはアミノ酸の性質によるものと主張しています。
短いRNAがミトコンドリア上にリクルートされるのはなぜ?
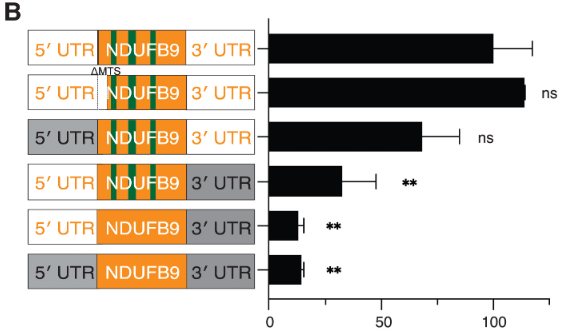
次に彼らは短いRNAについても変異体を色々試して、短いRNAのリクルートにはIntronと非翻訳領域(UTR)が重要であることを示しています。
下図では一番上が野生型の電子伝達系に関わる短いRNAで、上から2個目と3個目がMTS配列を欠損したMutantと5’側のUTRを欠損した変異体です。これらは局所翻訳に影響を与えませんでした。長いRNAと違ってMTSは局所翻訳には影響せず、そこからの挿入にのみ重要であるようです。
一方で3’側のUTR(上から4番目)やintron(5番目)を欠損した変異体では局所翻訳が有意に減少しています。
ここからハエでミトコンドリア上の翻訳に関係すると既報があったAKAP1に着目しています。AKAP1が欠損すると、短いRNA特異的に局所翻訳が低下することが分かり、またその制御にRNA binding domainが重要であることが分かりました。
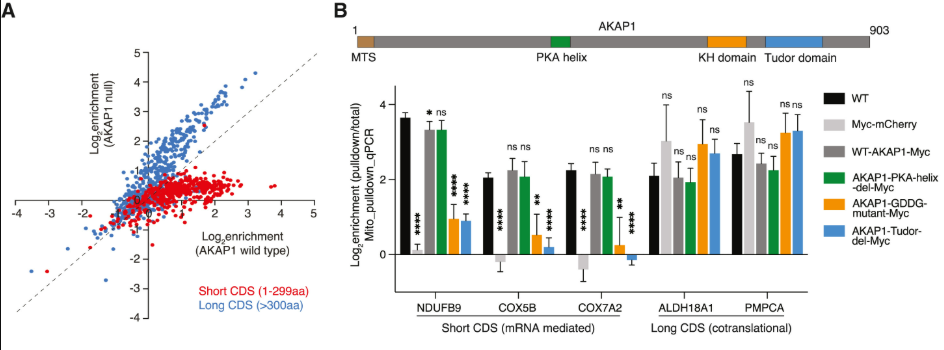
その後AKAP1が短いRNAに特異的に結合していることをRIP-qPCRで示して、その結合にintronと3’UTRが必要であることを示しています。
短いRNAの局所翻訳は進化的に新しい
最後にこの2つのメカニズムの局所翻訳が進化的にどの段階から現れるのかを調べています。
その結果長いRNAの局所翻訳は酵母から保存されているのに対して、短いRNAの局所翻訳は真核生物から出現する進化的に新しいメカニズムであることを示しています。
感想とQuestion
メカニズムはまだまだ分かってないことが多い印象ですが、ミトコンドリアにおける局所翻訳が証明されたのは大きな進展と思います。Ribosome profilingのプロらしい創意に満ちた実験系でした。
長いRNAのMTSから200塩基下流にある領域が重要であることは分かったのですが、それがなぜ重要かのさらなるメカニズムについては今後の研究が待たれますね。
あとは短いRNAでMTSを含まないミトコンドリア遺伝子もあるようですが、そういうタンパクはどうやって内膜に到達するのでしょうか。外膜への挿入であれば、分かるのですが電子伝達系のタンパクが内膜にどうやって挿入されるのか分かりませんでした。また時間があるときに既報を調べてみようと思います。
またDisucussionでも触れられているのですが、核内でSplicingされるはずのIntronが細胞質内での局在に影響をあたえる理由がよく分かりませんでした。Splicing後のRNAになにか修飾など痕跡が残るのでしょうか。
もし分かる人がいたらぜひ教えてください。


コメント