【本日紹介する論文はCC-BYになっており、画像や文章の商用引用も可能な論文です。】
https://s100.copyright.com/AppDispatchServlet?publisherName=ELS&contentID=S0092867425007354&orderBeanReset=true&orderSource=Phoenix
炎症が起きている組織や腫瘍組織では免疫細胞の浸潤以外にも低酸素や酸性化、高浸透圧などの様々な環境の変化が起きています。
これらの環境変化が免疫細胞における遺伝子発現に影響を与えていることは知られていますが、酸性化した組織において免疫細胞の炎症関連遺伝子の発現を制御するメカニズムは発見されていませんでした。
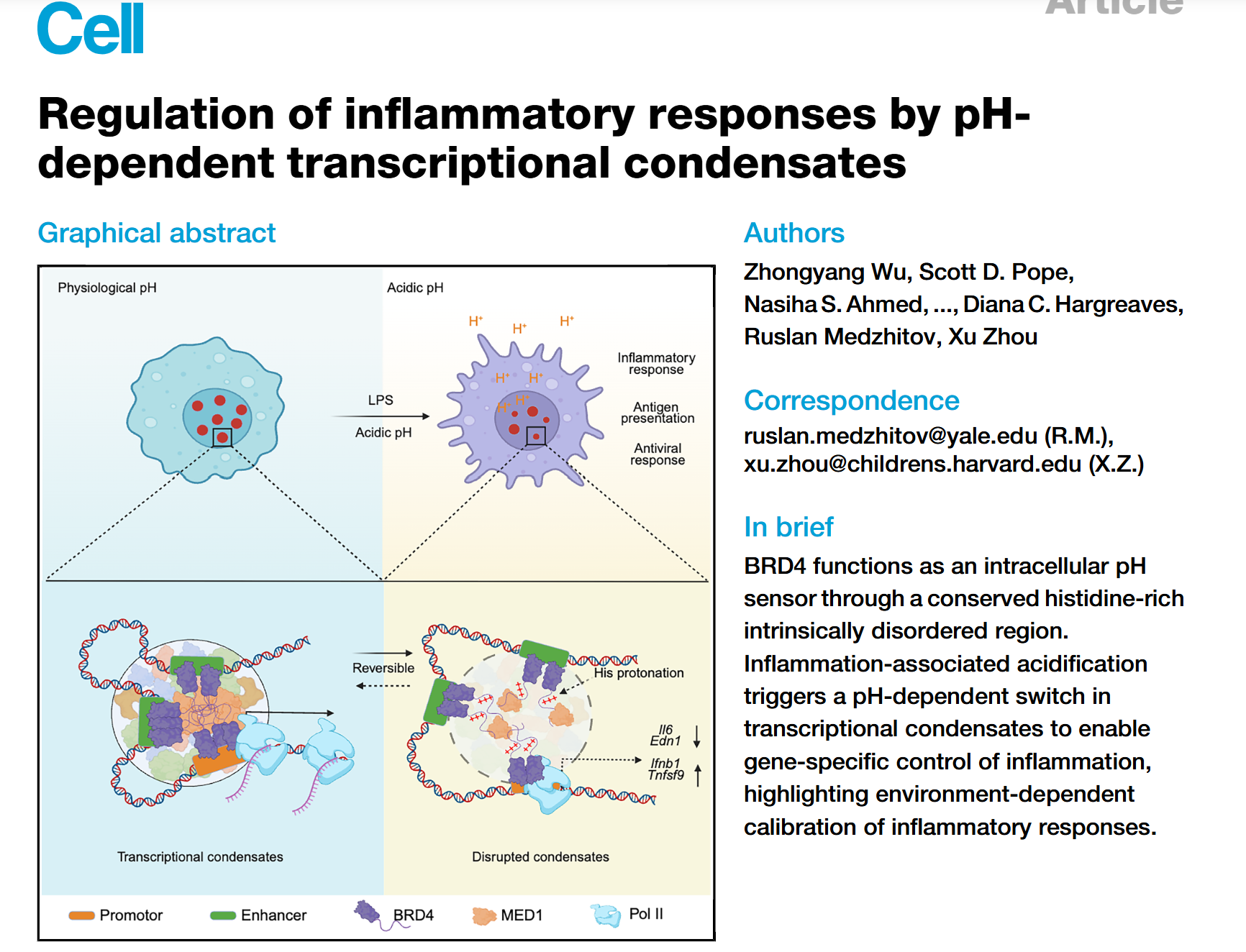
今回紹介する論文はハーバード大学のグループとRuslan Medzhitovによる共同研究の論文で、タイトルは「Regulation of inflammatory responses by pH-dependent transcriptional condensate」です。
(https://www.cell.com/cell/fulltext/S0092-8674(25)00735-4#figs5)
Ruslan MedzhitovはJanewayラボでポスドクをしていたTLR研究で有名な研究者ですが、最近は環境による免疫細胞の遺伝子発現変化について研究をおこなっています。
🧬炎症で酸性になると遺伝子のスイッチが変わる
彼らは初めに培地を酸性環境に変えたときに遺伝子発現が変化するかを調べました。そうすると遺伝子の種類によって酸性化によって発現が抑制される遺伝子と、増強される遺伝子があることが分かりました。
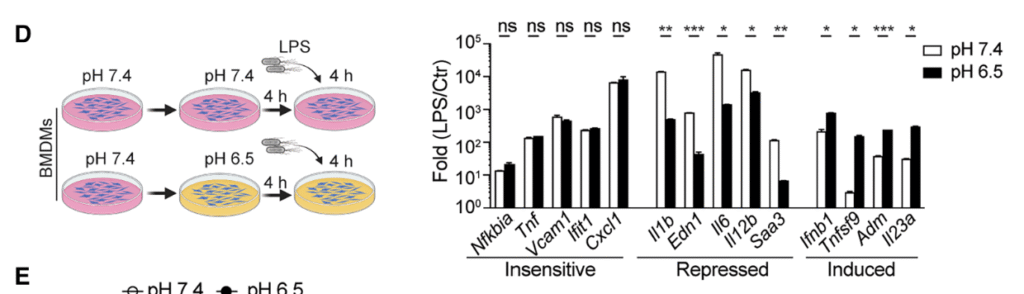
酸性化によってどのような遺伝子発現が変化するか調べる目的で、下記の4群でBulkのRNA-seqを行いました。
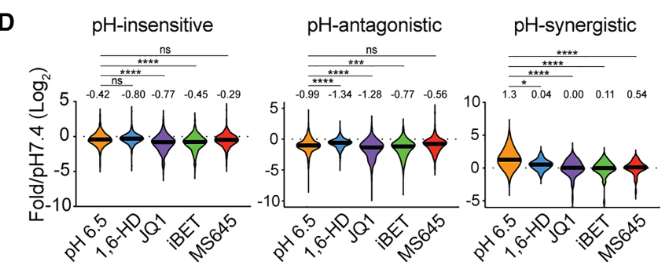
その結果、下記のように酸性化にSensitiveな遺伝子とそうでない遺伝子があることが分かりました。
Independent (pHIN): Nfkbib, Nfkbid, Nfkbie, Nfkbia and Nkfbizなど炎症関連の転写因子
Antagonistic (pHANTI): 炎症性サイトカイン (Il6, Il12a, Il12b, and Il18), ケモカイン (Ccl5, Ccl8, Ccl12, Cxcl9, Cxcl10, and Cxcl11)など
Synergistic (pHSYN): Adm, Ifnb1, Tnfsf9, Il23a, and Adora2b(抗ウイルス応答など)
TLR4下流の転写因子に差はない
TLR4にはアダプター分子であるMyd88とTRIFによる独立したSignal経路があり、その下流の転写因子のにNFκBとIRF3がありますが、これらのタンパク発現や局在には酸性化による変化を認めませんでした。
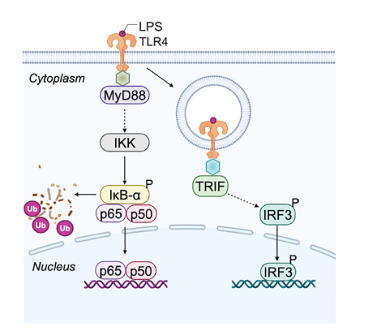

またEpigeneticな変化についてもATAC-seqやChip-seqで調べていますが、このpH依存的な遺伝子発現の変化を説明できるような変化は見つかりませんでした。
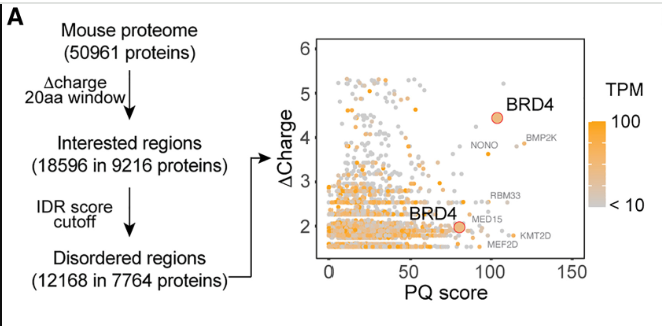
in silicoスクリーニングによるBRD4の同定
そこで酵母において酸性環境でヒスチジンの電荷が変化することで、Phase separationによりタンパクの局在や遺伝子発現が変化するという既報があったことから、似たようなメカニズムの可能性を調べました。(elife: SWI/SNF senses carbon starvation with a pH-sensitive low-complexity sequence;https://elifesciences.org/articles/70344)
まずはこの論文を参考に、マウスの蛋白で下記の条件を満たすタンパクをバイオインフォマティクスでスクリーニングしました。
・酸性条件で電荷が変化すること
・IDR領域があること
・プロリンやグルタミンが多いこと(酵母の論文を参考にしている)
その結果BRD4という転写に関連する遺伝子を同定しています。
実際に顕微鏡で調べてみるとマクロファージ核内のBRD4の凝集体が酸性条件で消失することを認めました。またPhase separationによるものかを1,6-hexanediol(1,6-HD)という試薬やFRAP(Fig.4I)というレーザーの実験で示しています。
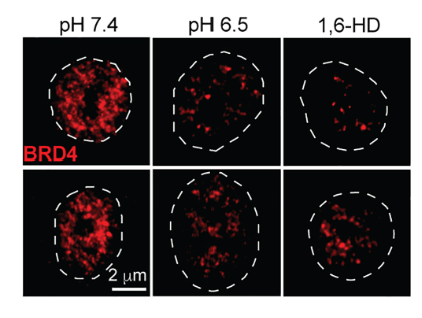
面白いことにこのタンパクによる凝集体はpHを酸性から中性に戻すと再び凝集体を形成します。HEK293TでLive imagingでもこのことを示しています(Video.S1)。

このBRD4が重要か調べる目的で、BRD4の阻害薬や1,6-HDを使用したところ酸性化と同様にpH-antagonisticな遺伝子の発現を抑制することが分かりました。(若干分かりにくいですが縦軸0の基準点から全体的に発現が低下しています)一方で、pH-synergisticな遺伝子についてはあまり影響を受けておらず、これらについてはBRD4 independentなメカニズムだろうと推測されています。
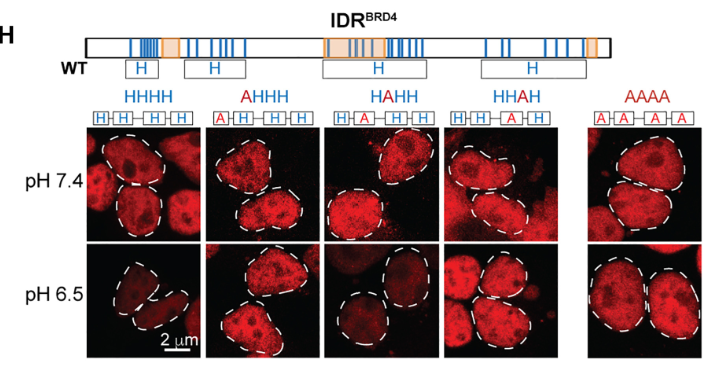
ここからBRD4のヒスチジンをアラニンに変えて、酸性化による電荷の変化を抑制することで、どの領域のヒスチジンが重要かを調べています。
BRD4依存の凝集体がエンハンサー領域を活性化する
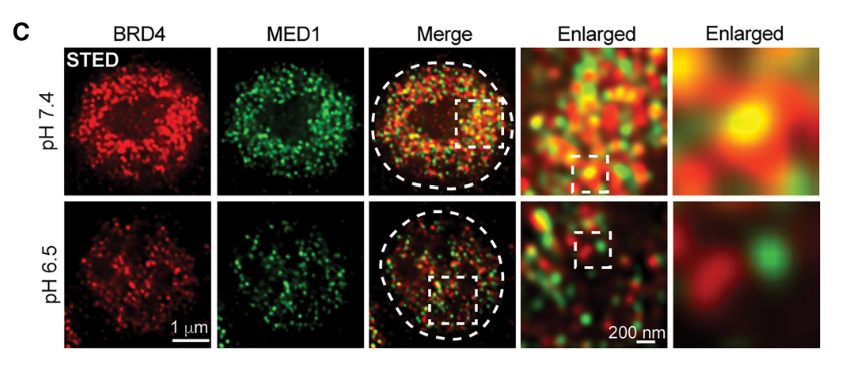
ここからさらになぜこの凝集体が転写活性化に重要かを調べています。2018年のScienceの論文(https://www.science.org/doi/10.1126/science.aar3958)で、BRD4とMED1がPhase separationによる凝集体に共局在し、転写活性化を制御すると報告されています。
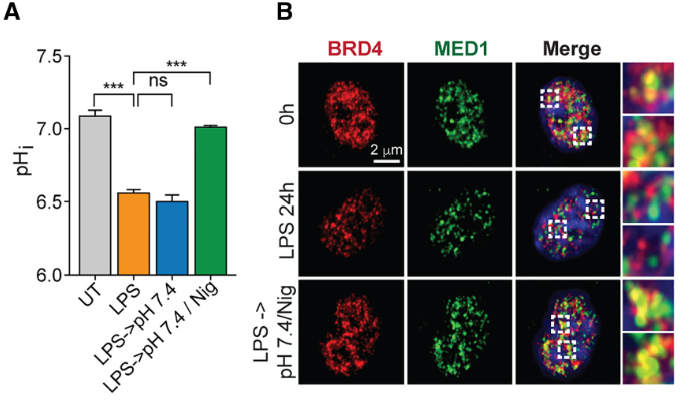
顕微鏡でみるとBRD4とMED1の共局在(黄色の信号)が酸性条件で減弱していることが分かりました。
以前のEpigeneticな解析でEnhancer領域でpH依存な変化があることが分かっていたことから、Enhancer領域へのBRD4の結合をChiPseqで調べました。その結果他の遺伝子と比較して、pHANTIでより結合していることが分かり、酸性条件下での遺伝子発現の特異性を示唆する結果となりました。
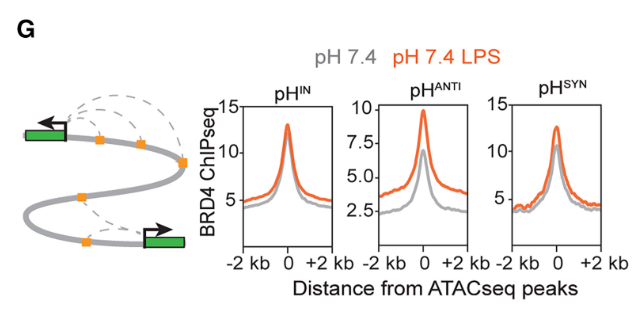
またこのエンハンサー領域の制御についてBRD4とともに、non-canonical BAF複合体を構成するBRD9が重要であることも示しています。なのでここまでの流れをまとめると
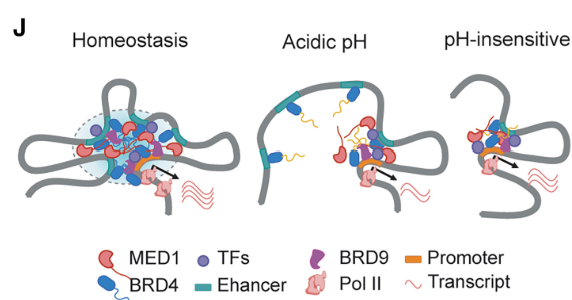
1.BRD4がpHセンサーとして機能し、BRD9と複合体を形成してMED1を含む転写凝縮体を作る
2.酸性pHになるとBRD4のヒスチジンがプロトン化され、BRD4-MED1凝縮体が減弱する
3.その結果BRD4とBRD9との結合も消失し、クロマチンリモデリングが行えなくなる。
4.結果として、pHANTI遺伝子(Il6, Il12bなど)のエンハンサー活性が低下し、転写が抑制される。
BRD4はGeneralなpHセンサーである
Nigericinというカリウムのイオノフォアと高濃度のKClを細胞外に入れることで、細胞内のH+とK+を交換することで、細胞内のpHを下げる実験を行うと、凝集体の形成が回復することも分かりました。
このあと腹腔内マクロファージを用いてin vivoの環境でも同様の現象がみられることや、BRD4の制御がLPSによる解糖系の活性化にも重要であることを示しています
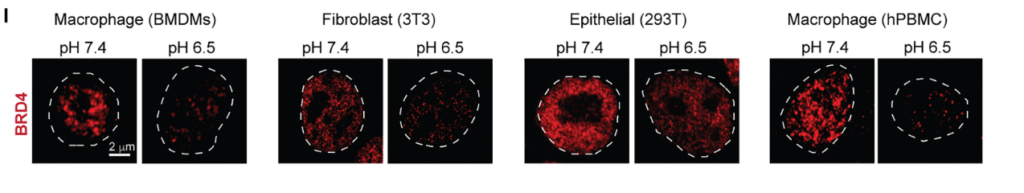
最後にこのBRD4がMacrophage特異的なものではなく、他の細胞種でも重要なpHセンサーであることを示しています。
感想
とても面白い論文で、新しいpHセンサーを同定できたのは大きなNoveltyだと思います。
個人的には下記のような点が気になりました。
Synergisticな遺伝子のGOでT細胞の分化に関わる遺伝子がとれていますが、実際にT細胞でどうなのか
シクロヘキシミド(CHX)の実験で酸性化と同じ表現がみられていますが(Fig.3B)、その後CHXが出てこないので関係あったのかが不明
Nigericinはカリウムの流出によりインフラマソームを活性化すると考えられていますが、インフラマソームの活性化にもカリウムとH+の交換によるpH変化の関与があるのか


コメント